GHGA GHGA Metadaten-Modell Version 0.4.0 veröffentlicht
- 17 Dez. 2021
- Florian Kraus
Im Dezember 2021 haben wir Version 0.4.0 des GHGA Metadaten-Modells veröffentlicht, das grundlegende Informationen für die Datenablage in GHGA liefert und eine solide Plattform für die weitere Entwicklung bietet.
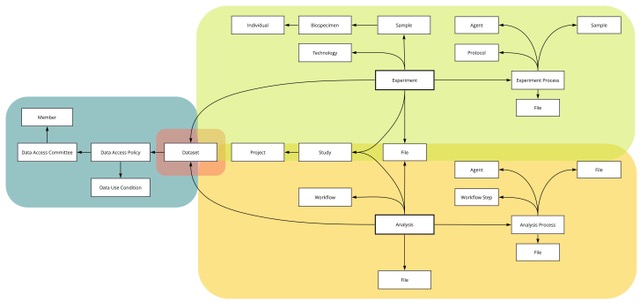
Der Metadaten-Workstream entwirft ein strukturiertes Modell für die im Deutschen Humangenom-Phenomarchiv (GHGA) gespeicherten Daten. Es handelt sich um eine gemeinsame Anstrengung der konzeptionellen und technischen Abteilungen des GHGA. Das Team besteht aus Expert:innen mit Erfahrung aus verschiedenen Bereichen, darunter Datenbanktechnologien, rechtliche Rahmenbedingungen, Community Standards und den FAIR Data Prinzipien, welche in die Implementierung des Konzepts der GHGA Metadaten einfließt.
Das Metadaten-Modell wurde in GHGAs GitHub Repository veröffentlicht. Dieses Kernmodell dient als Grundlage für weitere Entwicklungen unseres Metadaten-Modells. Die Erfassung wesentlicher Informationen, wie z.B. Spender:innen von Proben, Experimenten und die Analyse der Daten, erhöht bereits die Wiederverwendbarkeit der in GHGA hinterlegten Daten.
Darüber hinaus haben wir Ontologien identifiziert, die die zu erfassenden Daten definieren. Ontologien sind Definitionen, die Begriffe und deren Verhältnis zueinander beschreiben. Dies schafft eine Grundlage für gemeinsames Verständnis. Datenübermittler sehen so schon im Voraus, welche Informationen bereitgestellt werden müssen. Auch der Inhalt und die Bedeutung der von ihnen bereitgestellten Metadaten ist so eindeutig geklärt. Die Identifizierung weiterer Ontologien ist ein fortlaufender Prozess, der die Bemühungen des GHGA um die Erfüllung der W3C-Vision des "Web of Linked Data", des Semantic Web, unterstützen wird.
Das nun veröffentlichte Kern-Metadaten-Modell wird in naher Zukunft erweitert, um domänenspezifische Informationen zu erfassen. Derzeit arbeiten wir daran, die Anwendungsfälle der seltenen Krankheiten (Rare Diseases) und der Krebsforschung zu erfassen und gleichzeitig sicherzustellen, dass wir in der Lage sind, in Zukunft die gesamte Omics-Gemeinschaft zu bedienen.