GHGA Metadaten-Modell Version 0.9.1 veröffentlicht
- 31 März 2023
- Karoline Mauer
Mit der Veröffentlichung des GHGA Metadata Catalog haben wir Version 0.9.1 des GHGA Metadatenmodells und die entsprechende Version des GHGA Submission Spreadsheets veröffentlicht. Modell und Spreadsheet unterstützen die strukturierte Ablage nicht-personenbezogener Metadaten im GHGA Metadatenkatalog.
Der Metadaten-Workstream entwickelt das Modell für die in GHGA zu speichernden humanen Omics Daten. Das Team besteht aus Fachleuten aus verschiedenen Bereichen wie Datenbanktechnologien, rechtlichen Aspekten, Community-Standards und FAIR-Datenprinzipien. Gemeinsam definieren sie das Konzept der GHGA-Metadaten.
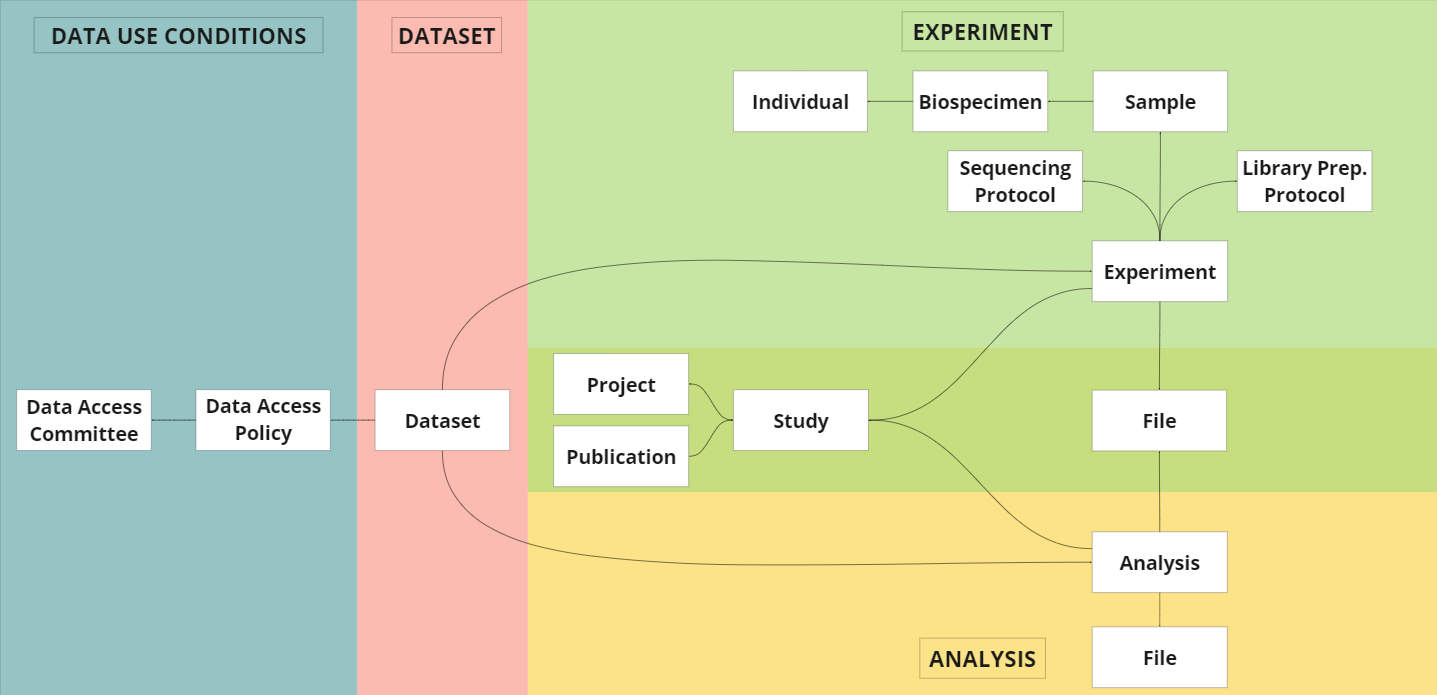
Das GHGA-Metadatenmodell wurde in GHGAs GitHub-Repository veröffentlicht. Die aktuelle Version des Modells ist eine Weiterentwicklung desGHGA Metadatenmodells, das erstmals im Dezember 2021 veröffentlicht wurde.
Die aktuelle Version enthält Listen kontrollierter Terminologien und standardisierter Ontologien und treibt damit die Harmonisierungs- und Standardisierungsbemühungen weiter voran, die ein wesentlicher Bestandteil der Ziele von GHGA sind. In enger Zusammenarbeit und Diskussion mit den künftigen Nutzer:innen des GHGA Metadata Catalogs und GHGA Archivs haben wir die Struktur des Modells verfeinert, die Verknüpfung zwischen den Entitäten verbessert und die Beschreibungen der Eigenschaften verbessert. Die Tabelle wurde umstrukturiert, um die Nutzer:innen während des Einreichungsprozesses zu unterstützen. Das Modell wird sich mit dem Beginn der GHGA Archivphase weiterentwickeln, um den Bedürfnissen verschiedener Communities und Anwendungsbereiche gerecht zu werden.
Aufbauend auf der früheren Zusammenarbeit mit dem ELSI-Workstream bei GHGA zeigt unser Modell, wie Omics- und Gesundheitsdaten in Übereinstimmung mit den allgemeinen Datenschutzbestimmungen (DSGVO) gespeichert und sicher zwischen deutschen Institutionen ausgetauscht werden können. Dabei wird das Recht des einzelnen Datenspenders auf Privatsphäre gewahrt und gleichzeitig hochwertige Metadaten erhoben, um Genomdaten in GHGA auffindbar und wiederverwendbar zu machen